内容提要
1 华南地区的原始森林
2 森林破坏的一般模式
3 香港森林的分布与特点
4 森林破坏之后自然恢复一段时间后的特征
5 森林能自己恢复吗?
6 人们希望恢复生态系统功能
7 人工恢复不如自然恢复吗?
8 我们正在做什么工作?
- 8.1 在草地上恢复森林
- 8.2 恢复浙江润楠、短序润楠和刨花润楠组成的次生林
- 8.3 恢复生态学实验的矛盾
9 结论与展望
致谢
大家好, 我叫张金龙,在香港嘉道理农场暨植物园植物保育部工作,主要负责植物标本馆和森林恢复样地的数据管理和分析。我是天津宝坻人,2006-2011年在中科院植物所读书,2013年开始在这里从事恢复生态学有关的工作。
一提到恢复生态学,很多人会想起种树。没错,我们的任务就是种树。从2013年到现在,我们已种下4万多棵树苗,大约300个树种,还有300多种灌木、草本等。另外,我们的苗圃为其他机构提供了几万棵本土树木的幼苗,种在香港的大屿山、大榄涌水塘附近。今天很荣幸有机会跟大家介绍一下这几年工作的收获以及相应的思考。
1 华南地区的原始森林
我们关注的是南亚热带和北热带地区的森林恢复,所以关注南方的森林比较多,特别是海南、广西、广东等地的森林。

图1. 海南尖峰岭的北热带季雨林 (摄影 张金龙)

图2. 广东黑石顶的亚热带常绿阔叶林 (摄影 张金龙)
这张照片是海南岛尖峰岭的原始森林。尖峰岭位于海南岛西南端,主体是北热带季雨林。这里所说的原始森林,已经不是人迹罕至了,实际上现在也很少有什么人迹罕至的地方了。之所以说这是原始森林,是因为这片林子过去没有经过大规模砍伐。那么在这样的森林,我们能看到什么?
- 森林分成几层,有乔木层、亚乔木层、灌木层、草本层等。
- 有很高大的树木,树冠层有40-50m高,胸径达1-2m甚至更大。
- 每一种树木,虽然有些个体数很多,有些很少,但是大都有大树,也有小树,也有幼苗。也就是说,这些种是在不断更新的。
- 森林当中存在着众多的藤本以及附生植物。
植物资源调查,如采集标本、做样方等,一般都是尽量选取这样人为干扰少的地方。因为这样的地段一般来说物种多样性非常高。去这样的地方调查,各种受保护的种、稀有物种也相对容易见到,甚至能发现新种。
华南地区的原始森林原本分布着很多种动物,如长臂猿、亚洲象、犀牛、野牛等,还有各种鸟类,爬行动物、两栖动物、昆虫等等,这些基本上都在保护区考察集里面列出来了。有一个问题,就是有些种,可能几十年前、几百年前比较多,最近变得十分稀少,甚至再也没有发现过了,这样的种一般也收录在考察集中。这样的物种其实有不少。为什么会这样呢?这很可能表明生态系统出现了问题,出现了明显的衰退。不仅在保护区外面如此,不少自然保护区里面也是如此。很多保护区的物种组成,特别是动物的物种组成,即使跟几十年前相比,也发生了一些变化。
比如尖峰岭现在最好的森林是中高海拔地区的。中高海拔的森林,从结构和科属的组成上看,与南岭以南各地,如广东鼎湖山、黑石顶、广西的十万大山以及越南北部的植物科属组成是非常相似的,有很多壳斗科、樟科的大树。另一个地方,广东封开黑石顶,属于南亚热带常绿阔叶林,在当地的山坡上,乔木层有些树种,如蕈树、黑叶锥等也能达到50m高度。这些地方的森林物种组成复杂,种类十分丰富。
有人问,你不是讲森林恢复吗?而且是讲嘉道理农场森林的恢复,这些地方的森林跟你们的森林恢复有啥关系?
当然有关系,这是因为,只有了解了附近区域最好的森林,才能推测要恢复的森林是什么样子,知道物种组成的差距,才能知道应该从什么角度,用什么方法去恢复本地的森林。邻近地区保存很好的森林,可作为森林恢复的模板。
为什么要恢复植被?很显然,是因为植被退化了,不能再提供我们需要的功能了,而森林可能难以自己恢复,所以需要我们去恢复。
在讲我们做过哪些尝试之前,我们先看看森林是怎么破坏的。
2 森林破坏的一般模式
这幅地图展现的是亚洲的人类足迹,我们发现,人类没有到达的地方确实是很少了。不仅如此,对于植被的破坏或者人为干扰,在农业早期,也就是几千年前就已经开始了。
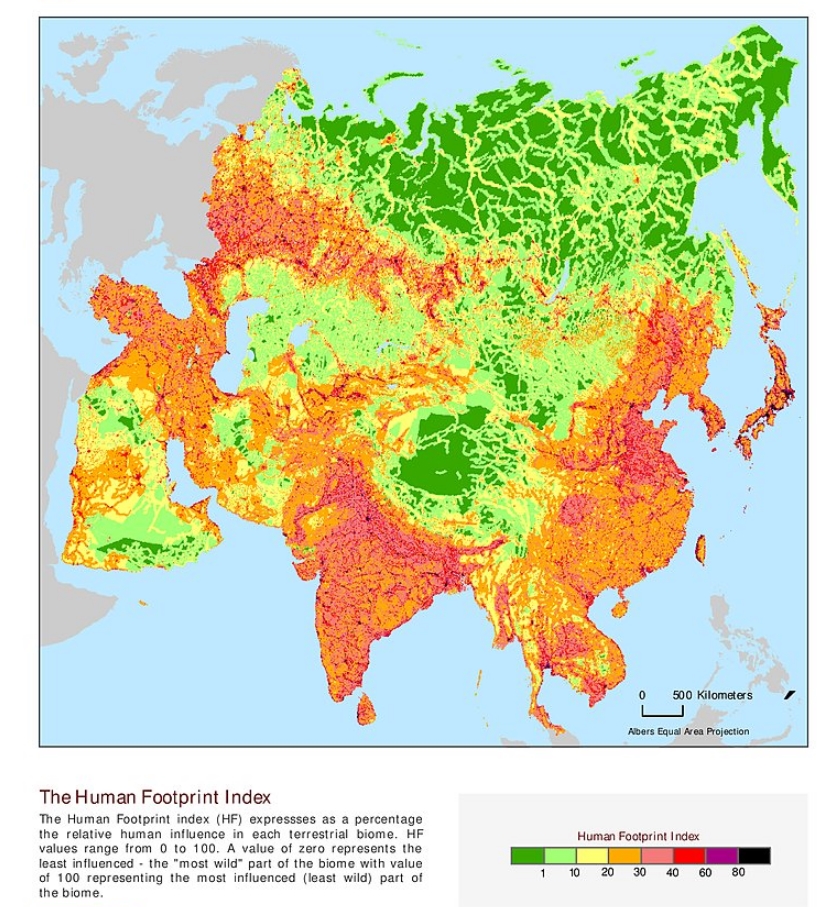
图3. 人类足迹 (来源 https://sedac.ciesin.columbia.edu/downloads/maps/wildareas-v2/wildareas-v2-human-footprint-geographic/hfp-asia.jpg)
地势平坦、低洼的地段,水源也丰富,适于人类居住和耕作,对于我国南部来说,这些地段也都是非常适合森林生态系统发育的。三四千年前,华南的森林比现在广布得多,当时环境可能也比现在温暖一些,那时,华南地区有亚洲象、犀牛、长臂猿等的分布。这些动物当中,很多已经在国内灭绝,或者现在的分布已经不在华南,而是分布在非常偏远或者非常狭窄的区域了。究其原因,除了人为猎杀,还有很大的原因就是毁林开荒,破坏了它们所依赖的生境。平原地区适于种植各种作物,特别是水稻。华南地区的水稻种植最晚在两千多年前的秦汉时期就已经开始了,也就是说森林破坏最晚从那时候就开始了。
多种人类活动,如农业、林业、城市化等都会导致植被退化。植被退化到一定程度,会有什么后果呢?我们以香港为例,看一下植被破坏后是怎样的。
3 香港森林的分布与特点
香港土地面积为1100多平方公里,分为香港岛、九龙和新界三部分,包括260多个海岛,其中面积最大的是大屿山。香港地形崎岖,主要是山地,最高峰是大帽山,海拔957m。年降水量约为2000毫米。降水在时间上非常不均匀,分为明显的雨季和旱季;空间上也非常不均匀,比如一些海岛,可能只有1800mm一年,而降水最多的是大帽山,年均达3000mm左右。年平均气温为22℃,因为位置靠南、靠海,平地的霜冻极少见,只有大帽山等几座“高山”上每年有几天有短暂霜冻。
.jpg&id=674800)
图4. 香港地形 (来源: http://map.ps123.net/china/17862.html)
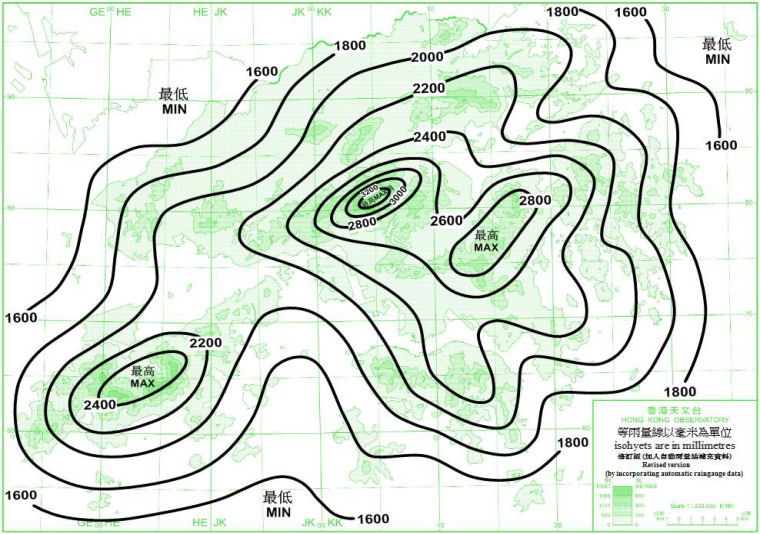
图5. 香港平均年雨量分布图 (1981-2010) (来源:香港天文台 https://www.hko.gov.hk/tc/wxinfo/climat/rfmap.htm)
这样的温度和降水非常适合季节性雨林的发育。在不少植被区划中,香港属于北热带,跟雷州半岛、海南岛、广西西南部以及越南北部属于同一个气候类型,因为地理距离很近,植物科属组成也是十分相似的,种类十分丰富。不过,由于几千年来,特别是最近几百年来,受到农业、频繁人为山火等的破坏,香港的植被破碎化极为严重。第二次世界大战之后,很多地段划为郊野公园,在近些年发育出多样性较低的次生林。
在此之前,如19世纪中叶,英国人登上这座岛屿的时候就感叹,这个地方非常贫瘠,属于“不毛之地”。
最晚在明代或者清代,香港大部分地区已经开辟为农田,而山上不便耕种的地段被开垦为茶山,虽然大部分茶山在清末便已经荒废,但不少地方留下了石头垒起来的矮墙,至今从照片上仍然可见。

图6. 香港大帽山(北坡),上部分为草地 (摄影 张金龙)
第二次世界大战时,日本占领香港达3年零8个月,香港的植被遭受进一步破坏,树木不仅被大量砍伐用作军事用途,普通人还需要大量的薪柴做饭、取暖等,使植被遭到非常严重的破坏。1945年的航拍照片清楚显示,在大帽山附近,森林覆盖率只有大约0.2%。残存的森林只分布在山顶附近的溪流中。这些地方石头太多,不适于耕种或者种茶,位置偏远,人也难以到达。
在其他地方,也有一些植被较好的地段,大约有几种情况:
- 村边的风水林。在华南地区,很多村附近有一小片树林。人们相信这小片树林能保佑村民平安,所以不会砍伐,偶尔也会种一些树,希望树林越来繁茂。这种树林保存了比较多低海拔,特别是具有热带性质的植物,如黄桐、白颜树、华润楠、荔枝、土沉香、显脉杜英等,偶尔能见到吊皮锥、红皮糙果茶等稀有种类。
- 非常陡峭的山体、瀑布等地段,或者非常偏远,交通不便的地段。在这些地区,有时生长有非常稀有的种类,如南华杜鹃、大屿八角、孔药楠、蓝树、观光木等。
虽然香港面积还没有内地很多县的面积大,人口也达到750万,但是仍然保存有2200种原生的维管植物。

图7. 香港新界大埔林村的风水林 (摄影 张金龙)
不过,由于香港的植被历史上受到的破坏很大,所以呈现出一些鲜明的特征:
4 森林自然恢复一段时间后的特征
- 物种组成改变:山顶和较高海拔的森林被草原代替。这种“草原”当然不是原生的,而是因为森林被破坏之后,原本在森林某些地段的阳生草本植物,扩散到它们适合的生境形成的,见于大帽山、八仙岭等新界北部各地,常见种类有鸭嘴草、芒、五节芒、鳞籽莎等。
- 物种多样性的改变:一般为物种数减少。森林被破坏之后,原本在某地区常见的种,可能就变得非常稀有,例如黄叶树,在华南地区是非常常见的种类,每公顷个体数最少应该在几百株以上,但是在香港却只有零星发现,个体数极少。一些广布的且在内地常见的树种,例如红锥,在香港目前只发现一个种群。
- 微环境的改变,物种更新困难:因为原本的森林生态系统被完全破坏,只有一些耐性非常好而且喜光的种类,如浙江润楠、短序润楠、白楸等,能够在草地上生长,逐渐恢复成结构简单的树林。而在山脊等非常暴露的位置或者频繁受到山火干扰的地段,一直是草地,很少有其他树种能够生长。
- 水土流失加剧。香港的降雨非常不均匀,有时24小时降雨量可达100-400mm,在这么强的降雨下,很多地方裸露的表土都被冲走了,由于淋溶作用的影响,土壤的营养物质极为匮乏。我们测量过嘉道理农场上山区森林恢复样地的土壤养分,其氮含量甚至低到仪器检测不到的水平。不难想象,在这样的地方,要进行森林恢复是十分困难的。
事实上,不同地段,因为破坏的程度不同,自然恢复的难度也不同,例如:
- 间伐:森林里面大树或名贵树种被砍。这种破坏对整个森林生态系统的影响比较小,但是对附生植物、野生动物以及树种的更新有较大影响。
- 皆伐:整片森林都被砍。乔木被破坏,但是灌木和乔木幼苗等大部分保存,种子库也未被破坏,如果有足够长的时间,有可能恢复为天然的次生林。
- 耕作:毁林开荒,林下层和种子库会被完全破坏。森林只能靠周围植被的种子和传播媒介更新。随着时间的推移,土壤层也被侵蚀和破坏。即使被森林生态系统包围,这种地段也是很难自然恢复的。
- 矿山:除了森林被完全破坏之外,矿山附近还伴随严重的土壤和水污染等,极难恢复。
除此之外,森林恢复能否取得成功,很大程度与还与恢复的策略,比如物种的选择、种植的方法等有关。
1970年以前,香港有不少大规模的植树计划,当局希望恢复森林,如在郊野公园范围里面种植了很多桉树、红胶木、白千层、台湾相思、马尾松等,这些树种有些已经种下去五六十年了,有些成功存活,但是它们生长的地段成了物种组成非常单一的树林,并不能算是真正的森林。有些种,如马尾松种植之后不久,就因为松材线虫、蚧壳虫等虫害、病害而大量枯死。现在,在香港几乎看不到马尾松了。这些人工林都没有按照传统的森林演替形成生物多样性更高的天然林。
5 森林能自己恢复吗?
森林恢复的重要性不言而喻,在健康的生态系统中,人能生活得也更舒服。但刚才也介绍了,香港有些地段的植被还可以,比如,大帽山的森林就是1945年那0.2%的森林恢复而来的,至今,大帽山山顶附近差不多也是植物物种组成也最为丰富的地段,我们去调查后也不断有新的发现,甚至发现了几十个香港新分布。那么问题来了,如果让森林自然恢复,不用太多人力物力,这不好吗?
我自己的答案是:在土壤被完全破坏,水土流失严重,自然恢复几乎是不可能的。主要原因如下:
第一,没有足够的种子能够自由传播到要恢复的地点,因为没有母树能够产生足够多的果实和种子,即使有点儿果实,果子狸、老鼠等动物天天惦记着,果实成熟之后,第一时间就被吃掉了。只有部分种类的种子能够在这种情况下存活。
第二,缺乏传播媒介。即使种子幸存,但是没有动物帮忙传播种子,种子就不能达到我们希望恢复的地方。有人说,有些种子是靠风或者重力或者水流传播,应该没问题。非常对,但是,这种情况下,因为传播方式的差异,有些种在天然恢复中比较成功,成为所谓的胜利者(winner species)。
第三,种子能不能萌发。假设种子能够传播到要恢复的地段,那么它们能顺利萌发并长出苗来吗?这涉及到种子所处地方的温度、光照、水分、土壤类型等。一般来说,种子都携带了母树给它的营养,很多种类能够供给几个星期甚至几个月的营养。问题是,不是所有的种类都能够萌发。究竟什么因素导致了种子不能萌发,不同种类有什么差别,这些都还需要研究。为了弄清楚原因,我们在农场内设置了实验,正在收集数据,希望能深入探讨。
第四,幼苗能不能生存和长大。幼苗能否长成幼树,也受到很多因素的影响,例如,水分、光照、疾病、同种的竞争、异种的竞争,动物取食、虫害、台风、旱灾、寒潮等等。这些因素中有些是相互重叠的。例如动物可能通过影响水分供给而影响幼苗的生存。有些动物能够非常准确地识别植物。比如,我们植树样地种了亚热带常绿阔叶林的常见种,石栎 ,但是野猪喜欢在它的根周围挖来挖去,我们样地里几乎每一棵石栎的根都被挖了。这些石栎的苗都没办法只能再次扎根,只能让从根部萌生了很多新的分枝,变成了灌木状。这样的树苗连生存都困难,也就失去了长成大树的机会。再比如,某种天牛非常喜欢在硬壳柯上产卵,幼虫将树皮环割,导致树苗枯死。另外,据重庆大学杨永川教授的研究,自然状态下,几千粒栲树的种子才能形成一棵幼苗。幼苗能不能生存,还跟土壤中的细菌和真菌有关,例如有些杜鹃必须要跟真菌形成菌根才能生存,豆科的绝大部分种类需要与固氮菌共生才能生存。在生境已经严重衰退的地方,这些有益的真菌和细菌是否存在,土壤退化后种类发生了哪些改变,都还不清楚,这方面的研究目前非常活跃。我们也正在跟北京林业大学等单位开展合作研究。

图8. 香港屯门青山的水土流失 (摄影 张金龙)
这张照片是香港屯门青山的水土流失情况,比大帽山的样地水土流失更严重。可以看到,这里土壤裸露,表土全无,有些地段只能见到鳞籽莎等。在这样的地方,森林的自然恢复几乎是不可能的。
6 人们希望恢复生态系统功能
有人要问,森林恢复不就是种树吗?这有什么好讲的。刨个坑,把树苗中下去,盖上土,再浇点儿水,就成了。
说得对,但是也不完全对,因为这是一般在公园、绿地里面种树的方法,而在亟需恢复的地段,种树要考虑的因素比这要复杂。而且,我们不仅仅是要把某一棵树种活,而是需要恢复整个森林生态系统的功能,包括固碳、涵养水源、防止水土流失等等。而且,我们还希望,在恢复的森林中,每个树种能够自我健康地更新,森林能跟天然森林一样,能够成为哺乳动物、鸟类的家园,能够具有和天然森林一样的景观文化价值。
7 人工恢复不如自然恢复吗?
恢复森林的多样性和全部生态系统功能是一种非常高的目标,目前来说,仍然是非常困难的。有人说,当代的最尖端的技术有几个,一个是太空探索;一个是对微观世界的探索,比如纳米技术、基因工程;另一个,就是在宏观尺度重建各种生态系统。前几个方向已经有各种进展,但是在宏观尺度重建生态系统目前还不知道怎样做到。如果人类能够成功恢复森林,就很可能能有效应对全球变化。但是,目前仍然有人怀疑人工恢复的成效。
两年以前,Science Advances上面有一篇论文,总结了全世界森林的恢复样地的恢复情况,最后认为人工恢复效果不如天然恢复。这篇论文发表之后马上引起了极大的反响。很快,就有生态学家指出,天然恢复的森林,一般来说,选取的样地是在森林容易自然恢复的地段;而人工恢复的样地,往往是矿山、或者植被严重退化的地段,这两种类型的样地,由于恢复的起点是不同的,不能直接简单比较。
为什么到现在还有生态学家觉得人工恢复没有自然恢复好呢?很大程度上是因为我们还不知道森林恢复的正确方法,不知道森林原本应该有什么物种组成和结构,不知道究竟需要把森林恢复成什么样。要恢复森林,我们首先要了解森林的复杂组成,这就是为什么要多了解邻近地区森林的物种组成和结构。
传统的森林演替模型说,如果一个地方,砍过之后,置之不理,大约能够在100-150年之内恢复到原始森林的状态。但是有研究发现,一个地区的森林能够恢复,取决于当地的物种库,如种子库或者幼苗是否存在,也取决于传播种子的动物等是否存在,取决于是否有持续的干扰等,并不是那么简单。研究发现,森林恢复过程中,生物量的恢复是容易的,也就是树冠层长得像原始森林一样大约需要80-100年的时间可能就够了,但物种组成恢复的中位数可能需要900-1000年。什么概念呢? 宋朝砍的森林,比如说,如果这一千年来没有经过太多的干扰,森林的物种组成才能和原始森林差不多。森林能恢复的前提是,所有的物种在生态系统中还存在。如果有些物种灭绝了,即使是本地灭绝,生态系统也可能就没有办法恢复到原始森林的状态了。
在这种情况下,也许要接受,进入人类世之后,某些生态系统再也没有办法恢复到以前的状态了。要将森林破坏后的植被恢复成健康的森林生态系统,提供必要的生态系统服务,支持野生动植物生存,需要从全新的角度考虑。
8 我们在做什么工作?
我们在大帽山有两种样地,一块是从2013年开始的,是从草地开始恢复的。另一块是从2018年开始的,是从天然恢复的润楠林开始。下面分别介绍:
8.1 在草地上恢复森林
明清时期,人们曾将大帽山较高海拔的地段开垦为茶园。清末,茶园逐渐荒废,植被退化,逐渐被草地所代替。过去,这些地段每隔几年或十几年就会受到一次山火的侵袭。
我们的森林恢复样地位于大帽山北坡,嘉道理农场暨植物园的最高处,海拔从500m到650m左右。我们每年都按照5m*5m的小样方种植几千棵树苗,分别设置为不同的实验处理。所以,不同年份种植的样方其实对应了不同的恢复生态学实验,尝试回答一些恢复生态学方面的问题。
所用树苗都是本土树种,目前,我们植物保育部有两个采集队,分别在香港的郊野公园采收种子,然后在苗圃培育树苗。
8.1.1 种子采集
在不同地点,采集种子后,编号并记录GPS信息。尽量在不同地点采集,以便提高所采集种子的遗传多样性,这样才能保证幼苗在种下之后,种群能够健康延续。有些稀有种由于很难采集到种子,也偶尔用采集来的枝条培育扦插苗。扦插苗的遗传多样性一般来说非常低,对于该物种的种群健康并不十分有利。不过,扦插苗所占的比例在我们的树苗中是非常小的。
8.1.2 播种和育苗
去掉果皮,清除果肉,播种到苗床。待幼苗几公分高时,移栽到塑料容器内。容器内装有沙土。这时候,每株幼苗都有自己的编号。在种植之前,需要测量幼苗的高度和基径。每一棵幼苗在种植之前,已经绑好了号牌。

图9. 嘉道理农场的本土树木苗圃 (摄影 张金龙)
8.1.3 种植
种植很简单,先用钢镐刨坑,深20-25cm,直径约30cm,将树苗根部的塑料袋去掉后再培好土。
参考CTFS (ForestGEO)大样地调查的方法,我们将样地划分成了20m20m 的样方,每个样方再划分成16个5m5m的小样方,向东为X,向北为Y。一般来说,每个5*5m样方是一种实验处理。
8.1.4 实验处理

图10. 嘉道理农场的森林恢复样地,蓝色为树木保护罩 (摄影 张金龙)
8.1.4.1 树木保护罩 (Treeguard)
有蓝色保护罩、遮阴网等几种。保护罩能明显提高幼苗的生存率并促进幼苗的生长。目前我们认为保护罩有以下几种作用:
- 降低光照强度,模拟幼苗最适宜光照
- 降低风速,降低干燥效应
- 提高幼苗的顶端优势
- 降低幼苗被草食动物(牛、赤麂、豪猪等)取食的风险
- 抑制杂草的竞争
8.1.4.2 防草垫
每棵幼苗周围铺设了防草垫,防草垫由椰子纤维压制,用来能抑制杂草的生长。不过,从长势来看,似乎防草垫对树苗生长的影响不大。
8.1.4.3 生物炭+堆肥 biochar + decompost
生物炭(biochar)是一种在特殊温度下烧制的炭,在土壤中能够长期稳定存在,也能够改善土壤的通透性和结构等,促进幼苗生长,与堆肥一起,用于改善土壤性质。
8.1.4.4 木屑 mulching
也就是将树枝粉碎,铺在树苗周围。这是一种园艺上常用的方法,可促进树木生长,抑制杂草。在土壤贫瘠地段,木屑分解后也能增加土壤肥力。
8.1.4.5 有机肥、化肥
为了增加土壤肥力,有些年份的实验中,我们施用有机肥,而部分样方在2013年施用了极少量的化学肥料。
8.1.4.6 修剪
在草地或者灌木丛中生长出的树木,可能因为没有足够的竞争者,植株往往在很早的时候就分枝,甚至出现多分枝结构,这样的树木在天然的森林里面原本应该是非常少的,但是在恢复样地却比较常见。为了能让种下去的树木能够像森林里面的树木一样,我们在树木长到5m左右时对其中一些个体的侧枝进行了修剪。这也是林业经营中常用的促进树木生长的方法。修剪能够在一定程度上提高林下层的光照,为后续的生物多样性强化种植创造条件。
8.1.4.7 间伐/稀疏化(thinning)
随着树苗长大,树冠层逐渐形成。如果树苗种类非常单一,个体密度又太高,将来长成的树林是非常不健康的,生物多样性自然提高十分困难。据以往经验,在树苗种植后三十年或者五十年之后,树种密集而单一的树林,植物种类仍然将十分贫乏。而且,如果在初始阶段种植得过密,个体之间的竞争会更为激烈,每一株都不能健康成长。这种树林的郁闭度非常高,林下非常阴暗,其他种的幼苗,甚至本种的幼苗也都很难生存。有人问,那如果不种那么密不行吗?答案是不行。根据经验,如果在一开始种植的时候树苗很稀疏,那么每个个体从保护罩中长出来后,生长会变得很慢,树冠层很难形成,森林恢复项目很可能会失败。为了形成连续的林冠层,目前流行的方法有三个:
- 宫胁(Miyawaki)法:这是日本生态学家宫胁昭提出的一种方法,也就是一开始种植密度非常高,例如每20m*20m种植几千棵幼苗,但是我们没有办法获得那么多的原生树种的幼苗,所以这种方法对我们来说并不适用。
- 保护罩法:用保护罩将每棵幼苗保护起来,去尽量模拟林下的条件,保证幼苗生存和生长。
- 抚育树法:先种一些能够快速生长的幼苗,作为抚育树(nurse crops),等到林冠层形成的时候,再砍掉一些,为后续种植创造合适的条件。
我们目前是将第二种和第三种方法结合,因此既要用保护罩,又要砍掉一部分抚育树。但什么样的树该砍,什么样的不该砍呢?有什么标准?目前仍然不清楚,只能说有一个原则,就是同一种植物,密度不宜太高。如果这些个体与要保留的某些个体有显著的竞争关系,则应该砍掉。
8.1.4.8 补种
为了提高样地的物种多样性,因此采用两步种植法。也就是在砍掉部分抚育树之后,再种植其他的:(1)树苗;(2)灌木;(3)草本;(4)附生植物等,以保证不同层的种类,在森林人工恢复的早期就存在,而不是在第一批树长大之后再自然出现。
8.1.5 数据收集
虽然有各种实验处理,但是我们仍然只能采集一些基本信息,比如幼苗的坐标和高度、基径等。要测量的指标,除了实验处理,还有:
- 位置:幼苗的X、Y坐标,然后转换为HK80坐标
- 物种:物种以及每个种的采集地点,一般在种子采集的时候,就已经鉴定到种了
- 基径:地面上5cm的直径
- 胸径:对于高度超过1.3m的植株,测量胸径
- 高度:也就是顶芽到地面的高度
- 冠幅:树冠的长轴和短轴
- 病虫害程度
- 整体表现:比如很好、一般或很差
除此之外,还要记录测量人、记录人等。数据收集好之后,输入Excel存档,以便进一步分析。
8.2 恢复浙江润楠、短序润楠和刨花润楠组成的次生林

图11. 以往的植树计划, 林下层很难恢复 (摄影 张金龙)

图12. 林下补充种植 (摄影 张金龙)
大帽山上天然恢复的森林,建群种多为浙江润楠、短序润楠、刨花润楠等,而其他亚热带建群种,如壳斗科、木兰科、金缕梅科、冬青科、山矾科、大戟科、桃金娘科植物都难以在短时间(几十年)内恢复。这些润楠林的种类单一,结构简单。一旦有严重的病虫害爆发,后果不堪设想。而且,目前已经发现一种由甲虫传播的真菌,导致大量润楠个体死亡。润楠死亡后,润楠的幼苗因为存在优先效应(priority effect),很可能又占据了林隙,长成大量幼苗和幼树,继续成为优势种。另外,天然恢复的润楠林, 以及有些植树计划种植的树林,林下过于阴暗,大部分树种的幼苗难以生长。不难想象,这些润楠林很难按照经典的森林演替模式演替,而是形成所谓的演替锁定(arrested succession)现象。这种现象是在植被破坏到一定程度后,物种无法自由传播和定植,优先效应导致优胜者(winner species)能够长期占据某一生境造成的。大帽山上天然恢复的润楠林以及以往种植的种类单一的树林,很可能进入了这样一种状态。因此,有必要开展实验研究,探讨怎样解除演替锁定。为此,我们尝试砍掉或者修剪了部分润楠个体,补种了更多种类,以提高多样性。
8.2.1 砍伐以及修剪
为了提高林下层的光照强度,大约要砍掉了一半左右的润楠个体,并对剩余个体的树冠进行修剪。
8.2.2 补种其他种类
补种什么种?我们补种了森林里面可能出现的: (1)其他常见树种;(2)灌木;(3)草本;(4)附生植物的幼苗。现在项目仍然在进行中。
8.2.3 监测什么?
当前主要是监测乔木幼苗和幼树的高度和基径等。对于其他植物,如附生种和草本植物,目前并没有统一的监测方法,我们正探讨如何进行如何对灌木、草本和附生植物的各种生长指标进行标准化。在环境因子方面,为了探讨影响每株幼苗生长的环境,每一棵幼苗我们都用鱼眼镜头拍摄了林冠覆盖度的全景照片。另外,我们也已经开始用无人机和激光雷达(Lidar)拍摄照片或者采集数据,尝试重建森林的三维结构,并据此推断每棵树苗的各种环境指标。
8.3 恢复生态学实验的矛盾
在恢复生态学中,样本量是非常重要的问题,各种恢复生态学中的实验处理,往往很难达到足够的样本量。要有足够的样本量,往往需要很大的面积,或者需要很多的个体。但是如果样地中“稀有种”的个体数远远高于在自然界中应有的数量,森林的物种组成必然又将严重偏离自然群落。由于每个种在生态系统中扮演的角色可能也是不同的,如果森林中稀有种的组成于显著偏离于天然群落,从长期来看,物种是否能稳定共存,还有很多不确定性。另外,这样的样地能够提供的生态系统服务与天然生态系统提供的服务是否一致也需要进一步评估。
9 结论与展望
综上,恢复生态学正处于转型的重要阶段,森林恢复是需要投入大量人力、物力的,凭借现在的知识和技能,目前人们还很难将森林恢复成与原始森林生态系统一样的水平。也就是说:
• 现有的森林恢复项目,在多样性上是远远不足的。
• 没有人知道森林恢复需要多长时间,目前只能尝试用各种方法,尽量在短时间恢复森林的物种组成以及结构。
• 对于生境破坏很严重的地段,只能凭经验选择用于恢复的树种。
• 森林恢复实验在样本量上一般难以达到统计上显著,在数据分析上也可能需要另辟蹊径。
• 以恢复多样性和生态系统结构和功能为目的森林恢复项目需要非常多的人力物力投入,在很大的空间尺度上是不容易做到的。
保护现存的天然植被,仍然是重中之重。努力理解和恢复复杂的森林生态系统,还有很长的路要走。要弄清楚这些关系,成功的恢复森林,就要走到森林里面去,去观察,去思考,去归纳总结。只有设身处地了解每种植物从种子到大树经历过什么困难、需要什么帮助,才能突破森林恢复的瓶颈。
致谢
最后,非常感谢各位的关注。感谢嘉道理农场暨植物园植物保育部主管Gunter Fischer博士分享自己的宝贵经验和想法。感谢每一位参与森林恢复项目的同事,感谢嘉道理农场暨植物园对项目的支持。



.jpg&id=674800)






